[연구동향] FastRNA: An efficient solution for PCA of single-cell RNA-sequencing data based on a batch-accounting count model
FastRNA: An efficient solution for PCA of single-cell RNA-sequencing data based on a batch-accounting count model
American Journal of Human Genetics, 2022


한범 교수(의과학과,공동 제1저자 및 교신저자) 이한빈 학생(의학과, 공동 제1저자 및 교신저자)
단일세포전사체 (Single-cell RNA sequencing, scRNA-seq) 자료는 C:\wizbox\project.webzine\관리자코딩-의대소식 표 있는 글.html 모든 유전자의 전사체량을 세포 수준에서 측정하는 기술로 종양, 자가면역질환 등의 정밀의료 발전에 크게 기여할 것으로 기대된다. 하지만 빠른 속도로 커지고 있는 데이터의 크기를 분석 알고리즘이 따라가지 못하고 있는 실정이다.
본 연구에서는 기존에 경제학과 역학에서 널리 사용된 조건부 회귀분석 (conditional regression analysis)과 특이값분해-고윳값분해 동등성 (Singular-value decomposition/Eigen decomposition equivalence)을 이용해 단일세포전사체 자료에서의 차원 축소 (Dimension reduction)와 변수 선택 (Feature selection)을 1000배 더 빠르고 효율적으로 수행할 수 있는 알고리즘인 FastRNA를 개발하였다. FastRNA를 이용하면 기존에 수 TB에 달하는 단일세포전사체 자료를 개인용 컴퓨터에서 수 분 이내에 분석할 수 있을 뿐 아니라 근사법이 아닌 수학적 동등성으로 최적화가 이뤄져 더 정확한 결과를 얻을 수 있다.
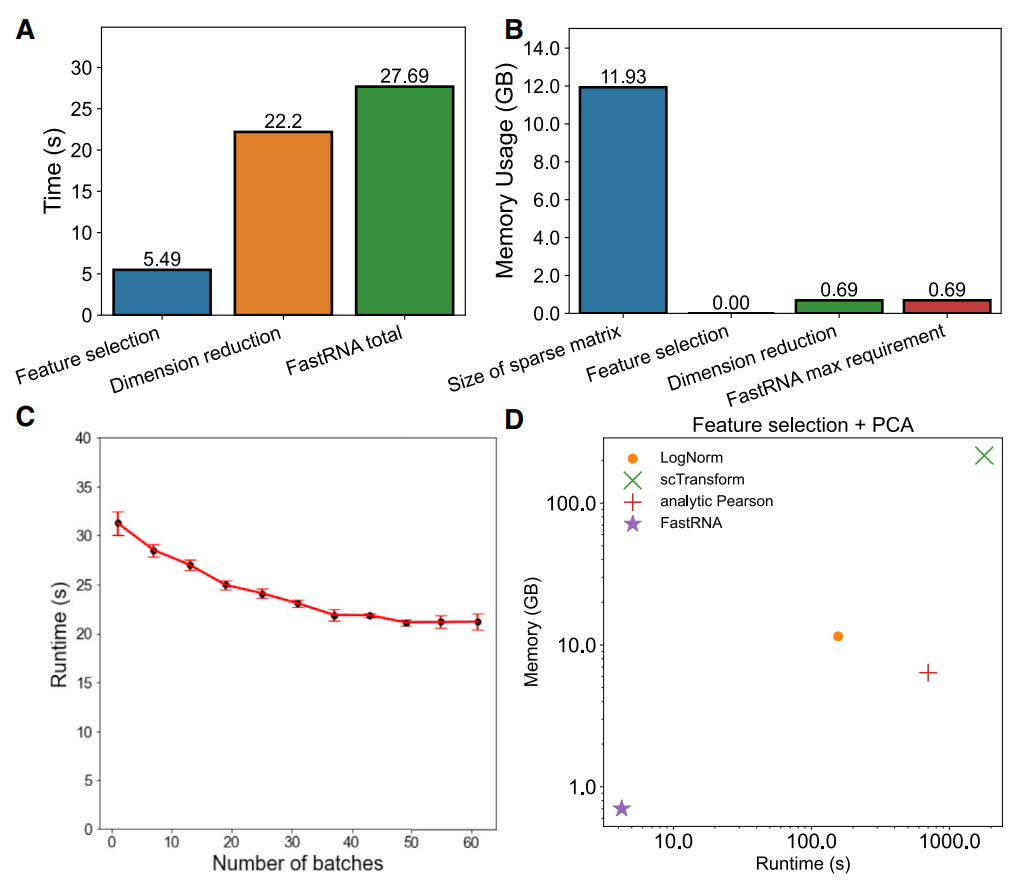
(A) FastRNA를 200만여 세포에 적용했을 때 걸리는 시간 (B) FastRNA를 200만여 세포에 적용했을 때 사용되는 메모리 (C) 공여자의 숫자에 따른 계산시간 (D) 20만여 개의 부분 샘플에 적용했을 때 다른 방법들과 시간 및 메모리 사용량 비교
논문링크 : https://www.sciencedirect.com/science/article/pii/S0002929722004116

